Unidad 7 Paquetes DiaThor y optimos.prime
Joaquín Cochero9
7.1 Paquete DiaThor
Autores: María Mercedes Nicolosi Gelis10, María Belén Sathicq11, Joaquín Cochero
7.1.1 ¿Qué hace?
El paquete calcula múltiples índices bióticos y ecológicos basados en diatomeas a partir de datos de abundancia en muestras ambientales.
7.1.2 ¿Cómo funciona?
El paquete obtiene las preferencias ecológicas de los taxones de diatomeas vinculandolos por nombre a las bases de datos de cada índice biótico y ecológico. Realiza una búsqueda exacta y luego heurística del nombre del taxón comparandola contra distintas Fuentes de información ecológica de las especies.
Una vez que encuentra la especie en cada alguna de las bases de datos de los distintos índices, realiza los cálculos de los índices en base a su abundancia en las muestras, y devuelve una tabla con los valores de cada índice por muestra ingresada.
7.1.3 ¡Manos a la obra!
Vamos a utilizar el set de datos del trabajo “Exploring the use of nuclear alterations, motility and ecological guilds in epipelic diatoms as biomonitoring tools for water quality improvement in urban impacted lowland streams”, de Nicolosi Gelis et al. (2020) (María Mercedes Nicolosi Gelis et al. 2020) publicado en Ecological Indicators y disponible en el Repositorio Institucional CONICET Digital.
7.1.3.1 Descargar los datos
Descargar los datos de ejemplo desde el repositorio del curso y guardar el archivo en el Directorio de Trabajo del Proyecto que creamos para esta Unidad (ver cómo hacerlo en la Unidad 1).
Datos de ejemplo aquí: https://github.com/Limno-con-R/CILCAL2023/blob/main/datasets/sampleData_diaThor.csv
7.1.3.2 Instalar el paquete
Instalaremos el paquete diaThor(M. M. Nicolosi Gelis et al. 2022) como se indica en la Unidad 1 y los cargaremos a nuestro espacio de trabajo actual:
library(diathor)7.1.3.3 Cargar y previsualizar nuestro archivo de datos
Cargaremos nuestro archivo de ejemplo (sampleData_diaThor.csv) a un dataframe llamado “base”:
base <- read.csv("sampleData_diaThor.csv") #Corroborar que el nombre del archivo coincida con el que vamos a utilizar, y que se encuentre en la carpeta de nuestro proyectoVeamos brevemente cómo está organizada la información de nuestras especies:
str(base)base es un objeto data.frame con 164 obs. o filas (especies) y 110 variables o columnas (muestras).
Nuestra planilla de datos además contiene una columna obligatoria llamada “species”, la primera, adonde debremos ingresar los nombres de las especies lo mejor que podamos, sin datos sobre autorías de las mismas.
Ejemplo de cómo llamar a las especies:
Hippodonta capitata ————> (ASI SI! ✔️)
Hippodonta capitata (Ehrenb.) Lange-Bert., Metzeltin and Witkowski 1996 ————> (ASI NO! 🚫)
Veamos las primeras filas y las primeras columnas:
head(base[1:10, 1:5])## X species S1_LI1_DAY0 S1_LI2_DAY0 S1_LI3_DAY0
## 1 1 Achnanthidium minutissimum 0 0 0
## 2 2 Achnanthes exigua var. exigua 0 0 0
## 3 3 Amphora libyca 0 0 0
## 4 4 Anomoeoneis sphaerophora 0 0 0
## 5 5 Craticula ambigua 0 0 1
## 6 6 Caloneis bacillum 0 0 0Cada columna representa a una muestra.
Debe contener un nombre de muestra (por ejemplo, se llaman S1_LI1_DAY0, S1_LI2_DAY0, etc.).
NOTA: Los valores en las celdas pueden ser valores de densidad absoluta (como es en este ejemplo) o abundancia relativa. Por defecto, diaThor asume que las densidades son absolutas; si queremos cambiar esta opción, debemos pasar el parámetro isRelAb=FALSE a isRelAb=TRUE cuando usamos las funciones.
7.1.3.4 ¡A la magia!
Vamos a ejecutar la función diaThorAll() , que se encargará de realizar todos los análisis que puede realizar el paquete, una atrás de otra. Y dispondremos de los resultados en un único objeto de tipo data.frame, que aquí lo llamaremos resultados.
Más adelante veremos que podemos solicitar cosas específicas si sólo necesitamos un tipo de resultado, para ahorrar tiempo y reducir la cantidad de salidas.
resultados <- diaThorAll(base)El paquete nos irá mostrando información en la consola de RStudio, y nos solicitará que le indiquemos una carpeta adonde guardar todas las posibles salidas que vaya generando. Exploraremos estas salidas luego para tratar de mejorar los resultados.
[1] "Select Results folder"
Seleccionemos un directorio (preferentemente vacío!) para guardar las salidas, y el paquete continuará reconociendo las especies, y buscando información ecológica sobre ellas para calcular los distintos índices bióticos.
Aclaración: Si estamos trabajando en sistema operativo Linux, es posible que este cuadro de dialogo no aparezca en la pantalla y obtengamos un error en la consola. Para evitar esto, podemos manualmente indicar el directorio adonde se guardaran los resultaos como un parámetro de la función diaThorAll, de la siguiente manera:
resultados <- diaThorAll(base, resultsPath="/Resultados") #Por ejemplo, aquí debemos haber generado una subcarpeta llamada 'Resultados'7.1.3.5 Los resultados
Si todo ha salido bien, diaThor debería haber calculado todos los índices posibles.
Cuando los índices bióticos se publican, no necesariamente se le asigna un valor ecológico a todas las especies de diatomeas (hay unas 200.000 especies!). Hay índices que sólo contemplan menos de 300 especies (ejemplos: IDP, SPEAR o los de Lobo et al.), y otros incluyen más de 6000 (ejemplo: IPS).
En la consola veremos que porcentaje del listado de especies que ingresamos fue efectivamente utilizado para calcular cada índice. Por ejemplo -> “Taxa recognized to be used in DISP index: 22.6 %”, nos indica que de nuestras 164 especies, sólo utilizó 22.6% para calcular el índice DISP, mientras que dejó afuera el 77.4%. Es importante entonces delimitar qué índices fueron calculados sobre una base sólida de nuestros datos al momento de interpretar los resultados.
El final de la secuencia de cálculo en la consola está delimitado por la exportación de gráficos ([1] “Plots exported!”).
En el objeto resultados veremos una tabla completa con todos los resultados obtenidos.
str(resultados)Es un objeto data.table con 109 obs. (nuestras muestras, filas) y 124 vars. (los índices calculados para cada muestra).
Veamos los resultados completos!
View(resultados)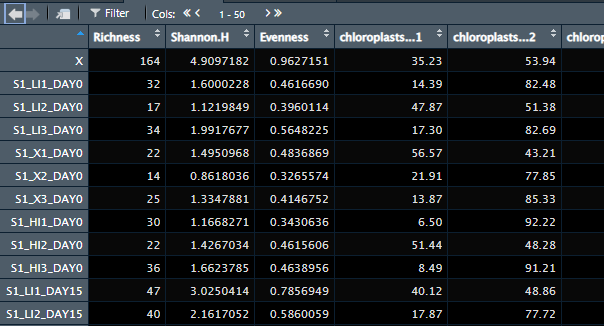
Figura 7.1. Primeras filas y columnas de la tabla de resultados de DiaThor
Veremos que cada muestra (fila) tiene sus valores de diversidad (Richness, Shannon.H, Evenness), porcentajes de número y forma de cloroplastos (chorloplasts, shape.chloroplasts), porcentaje de diatomeas de cada clase de tamaño (size.class), porcentaje de diatomeas de cada gremio ecológico (guilds), los valores de cada índice de Van Dam (VD.Salinity, VD.NHet, VD.Oxygen, VD.Saprobity, VD.Aero, VD.Trophic) y luego todos los índices bióticos.
Luego de cada uno de los índices, se encuentra una columna que indica cuántas especies no fueron determinadas en cada muestra (terminan en “.Indet”) y cuantas fueron usadas (terminan en “.Taxa.Used”).
7.1.3.6 La sintonía fina: veamos las salidas!
Una vez que el paquete termine el análisis, vaya a la carpeta adonde se guardaron los resultados. Se encontrará con varios archivos:
Diato_results - Results.csv: Este archivo contiene la tabla principal de resultados, con cada muestra (fila) y sus valores de los diversos índices que si se pudieron calcular (columnas).
Plots.pdf: Se incluyen los gráficos de cada uno de los índices bióticos calculados (eje X) en cada uno de las muestras analizadas (eje Y) para poder visualizarlo. Son realizados con los mismos datos que se encuentran en la tabla de resultados.
Taxa excluded y Taxa included: Es MUY importante que se revisen estos dos archivos. En estos archivos veremos que taxa de nuestra lista de especies original fueron incluídos o excluídos en el cálculo de cada uno de los índices. También nos ayudará a reconocer potenciales errores de tipeo o de nombramiento de especies.
VanDam Taxa used.txt: En particular, los valores ecológicos de Van Dam son separados en un archivo de texto al resto de los resultados, porque suelen ser muy amplios. Aquí se indica en cada una de nuestras muestras los taxones que fueron incluídos en el cálculo de cada una de las tolerancias (salinidad, N-heterotrofía, requisitos de oxígeno, saprobiedad, humedad, estado trófico).
Es muy importante que antes de reportar los resultados del análisis, se revisen cuidadosamente las salidas del paquete. Nos pueden indicar que hay especies con el nombre mal escrito, o que para el cálculo de algún índice en particular se utilizó poca cantidad de especies presentes, cuestionando así la precisión del mismo.
7.1.3.7 Algunas aclaraciones relevantes:
Sobre el reconocimiento de los nombres de las especies:
DiaThor realiza dos búsquedas automáticamente de cada especie en nuestro archivo.
Primero realiza una búsqueda exacta de cada especie comparandola contra todas las especies utilizadas en todos los índices bióticos y ecológicos.
Aquellas especies que no son encontradas por este medio, las separa y las vuelve a buscar automáticamente por una búsqueda heurística, para tratar de contemplar aquellos errores en la escritura de los nombres (Quien no escribió incorrectamente ‘Nitzschia fasciculata’ alguna vez, que tire la primer piedra!).
Este tipo de búsqueda permite que haya un número determinado de caracteres distintos entre dos palabras para considerarlas iguales. Por defecto, este valor en las funciones de diaThor es igual a 2. Por ejemplo, la búsqueda heurística encontraría a la especie ‘Nitzschia fasciculata’ aunque la hubieramos escrito como ‘Nitzschia fasiculata’, porque hay sólo 1 caracter distinto. Este valor de distancia se puede cambiar con el parámetromaxDistTaxa.¡Cuidado! Mientras más alto el valor de este parámetro, más chances de encontrar la especie que buscamos, pero más posibilidad de que confunda nuestra especie con otra de nombre similar, y se incrementa notablemente el tiempo que tarda en buscarlas.
Las especies que tienen categorías subespecíficas (variedades y/o formas) se deben indicar con “var.” y “fo.”.
- Ejemplo: Achnanthes exigua var. exigua” o “Gomphonema parvulum var. parvulum fo. parvulum”. El texto no busca formatos, no es necesario poner las especies en itálicas o subrayadas.
El paquete va a tratar de buscarlas de cualquier manera aunque no contengan estos acrónimos, pero las búsquedas son más precisas y rápidas cuando estan nombradas así.
- Ejemplo: Achnanthes exigua var. exigua” o “Gomphonema parvulum var. parvulum fo. parvulum”. El texto no busca formatos, no es necesario poner las especies en itálicas o subrayadas.
Preguntas frecuentes
¿Hay problemas encontrando el archivo de entrada CSV?
La consola nos indica este error:
*cannot open file 'nuestro_archivo.csv': No such file or directorySi corremos la función
diaThorAll()directamente, sin agregar argumentos o parámetros, un cuadro de diálogo nos pedirá que seleccionemos el archivo con nuestros datos.
resultados <- diaThorAll() #sin argumentos- ¿Nuestros datos están en abundancia relativa?
Hay que indicarselo, cambiando el argumentoisRelAbaTRUE.
resultados <- diaThorAll(isRelAb=TRUE)- ¿No encuentra las especies, y quiero hacer una búsqueda heurística más amplia?
Cambiemos el argumentomaxDistTaxa.
resultados <- diaThorAll(maxDistTaxa=4) #Ahora buscará nombres de taxa en las bases de datos internas con hasta cuatro caracteres de diferencia al ingresado en nuestro CSV.- ¿Y si quiero sólo calcular un índice, en vez de todo? Así es más rápido.
Podemos usar funciones para cada índice biótico o ecológico.
#Primero, cargamos nuestros datos, como siempre
base <- read.csv("sampleData_diaThor.csv") #Corroborar que el nombre del archivo coincida con el que vamos a utilizar, y que se encuentre en la carpeta de nuestro proyecto
#Luego, necesitamos que diaThor reconozca las especies por su nombre. Para esto existe la función diat_loadData()
#A esta función se le pueden pasar los mismos argumentos que a diaThorAll(). Es decir, podemos restringir la distancia heurística con 'maxDistTaxa', o indicar si los datos están expresados en abundancia relativa con 'isRelAb'
resultados <- diat_loadData(base, isRelAb=TRUE, maxDistTaxa = 2) #Ejemplo usando abundancias relativas y la distancia heurística por defecto = 2
#Deberemos indicar una carpeta para los resultados, en el cuadro de diálogo
#Y luego, por ejemplo, podemos calcular sólo el índice IDP
resultadosIDP <- diat_idp(resultados) #Grabamos los resultados en el objeto resultados IDP
#o el índice IPS
resultadosIPS <- diat_ips(resultados) #Grabamos los resultados en el objeto resultados IPS
#Hay funciones separadas para todos los índices del paquete; en la documentación se indican cuales, y se van agregando con las distintas actualizaciones.7.1.4 Fuentes de información ecológica de las especies
La información morfológica se busca en la base de datos del proyecto ‘Diat.Barcode’:
- Rimet F., Gusev E., Kahlert M., Kelly M., Kulikovskiy M., Maltsev Y., Mann D., Pfannkuchen M., Trobajo R., Vasselon V., Zimmermann J., Bouchez A., 2019. Diat.barcode, an open-access curated barcode library for diatoms. Scientific Reports. https://www.nature.com/articles/s41598-019-51500-6
La clasificación de las especies por su clase de tamaño se obtiene de:
- Rimet F. & Bouchez A., 2012. Life-forms, cell-sizes and ecological guilds of diatoms in European rivers. Knowledge and management of aquatic ecosystems, 406: 1-14. https://www.kmae-journal.org/articles/kmae/abs/2012/03/kmae120025/kmae120025.html
La clasificación de las especies en gremios ecológicos se obtiene de:
- Rimet F. & Bouchez A., 2012. Life-forms, cell-sizes and ecological guilds of diatoms in European rivers. Knowledge and management of aquatic ecosystems, 406: 1-14. https://www.kmae-journal.org/articles/kmae/abs/2012/03/kmae120025/kmae120025.html
La clasificación combinada de gremios y clases de tamaño (más nueva) se obtiene de:
- B-Béres, V., Török, P., Kókai, Z., Lukács, Á., Enikő, T., Tóthmérész, B., & Bácsi, I. (2017). Ecological background of diatom functional groups: Comparability of classification systems. Ecological Indicators, 82, 183-188. https://www.sciencedirect.com/science/article/abs/pii/S1470160X1730420X
Preferencias ecológicas para la clasificación de Van Dam se obtienen de:
- Van Dam, H., Mertens, A., & Sinkeldam, J. (1994). A coded checklist and ecological indicator values of freshwater diatoms from the Netherlands. Netherland Journal of Aquatic Ecology, 28(1), 117-133.
Los índices de diversidad (Shannon H’) se calcula usando internamente el paquete vegan (Oksanen et al. 2022):
- Shannon, C. E., and Weaver, W. (1949). ‘The Mathematical Theory of Communication.’ (University of Illinios Press: Urbana, IL, USA.)
Los valores de tolerancia y valencia de cada especie para cada índice biótico se obtiene de sus fuentes publicadas originales:
IPS: Coste, M. (1982). Étude des méthodes biologiques d’appréciation quantitative de la qualité des eaux. Rapport Cemagref QE Lyon-AF Bassin Rhône Méditerranée Corse.
TDI: Kelly, M. G., & Whitton, B. A. (1995). The trophic diatom index: a new index for monitoring eutrophication in rivers. Journal of Applied Phycology, 7(4), 433-444.
IDP: Gómez, N., & Licursi, M. (2001). The Pampean Diatom Index (IDP) for assessment of rivers and streams in Argentina. Aquatic Ecology, 35(2), 173-181.
DES: Descy, J. P. 1979. A new approach to water quality estimation using diatom. Beih. Nov Hedw. 64:305-323
EPID: Dell’Uomo, A. (1996). Assessment of water quality of an Apennine river as a pilot study for diatom-based monitoring of Italian watercourses. Use of algae for monitoring rivers, 65-72.
IDAP: Prygiel, J., & Coste, M. (1993). The assessment of water quality in the Artois-Picardie water basin (France) by the use of diatom indices. Hydrobiologia, 269(1), 343-349.
ID-CH: Hürlimann J., Niederhauser P. 2007: Méthodes d’analyse et d’appréciation des cours d’eau. Diatomées Niveau R (région). État de l’environnement n° 0740. Office fédéral de l’environnement, Berne. 132 p
ILM: Leclercq, L., & Maquet, B. (1987). Deux nouveaux indices diatomique et de qualité chimique des eaux courantes. Comparaison avec différents indices existants. Cahier de Biology Marine, 28, 303-310.
LOBO:
Lobo, E. A., Callegaro, V. L. M., & Bender, E. P. (2002). Utilização de algas diatomáceas epilíticas como indicadoras da qualidade da água em rios e arroios da Região Hidrográfica do Guaíba, RS, Brasil. Edunisc.
Lobo, E. A., Bes, D., Tudesque, L., & Ector, L. (2004). Water quality assessment of the Pardinho River, RS, Brazil, using epilithic diatom assemblages and faecal coliforms as biological indicators. Vie et Milieu, 54(2-3), 115-126.
SLA: Sládeček, V. (1986). Diatoms as indicators of organic pollution. Acta hydrochimica et hydrobiologica, 14(5), 555-566.
SPEAR(herbicides): Wood, R. J., Mitrovic, S. M., Lim, R. P., Warne, M. S. J., Dunlop, J., & Kefford, B. J. (2019). Benthic diatoms as indicators of herbicide toxicity in rivers–A new SPEcies At Risk (SPEARherbicides) index. Ecological Indicators, 99, 203-213.
PBIDW: Castro-Roa, D., & Pinilla-Agudelo, G. (2014). Periphytic diatom index for assessing the ecological quality of the Colombian Andean urban wetlands of Bogotá. Limnetica, 33(2), 297-312.
DISP: Stenger-Kovács, C., Körmendi, K., Lengyel, E., Abonyi, A., Hajnal, É., Szabó, B., Buczkó, K. & Padisák, J. (2018). Expanding the trait-based concept of benthic diatoms: Development of trait-and species-based indices for conductivity as the master variable of ecological status in continental saline lakes. Ecological Indicators, 95, 63-74.
7.2 Paquete optimos.prime
Autores: Belén Sathicq, María Mercedes Nicolosi Gelis, Joaquín Cochero
7.2.1 ¿Qué hace?
El paquete calcula los valores óptimos y rangos de tolerancia ecológicos de los taxones a las variables ambientales que proveamos.
7.2.2 ¿Cómo lo hace?
El enfoque más común utilizado para el cálculo de los óptimos y los rangos de tolerancia ecológicos es calcular la media ponderada (Birks et al. 1990; Potapova y Charles, 2003).
La media ponderada \(u_{k}\) de las estimaciones de los óptimos de las especies se calcula como:
\[ u_{k} =\sum_{i=1}^{n} y_{ik}x_{i}/\sum_{i=1}^{n}y_{ik} \]
y la tolerancia, o desviación estándar ponderada \(t_{k}\), se calcula como:
\[ t_{k} = \sqrt{\frac{\sum_{i=1}^{n} y_{ik} (x_{i} - u_{k})^2}{\sum_{i=1}^{n} y_{ik}}} \]
adonde \(y_{ik}\) es la abundancia relativa de la especie \(k\) en la muestra \(i\); \(x_{i}\) es el valor log10 del parámetro ambiental en la muestra \(i\); y \(n\) es el número total de muestras del conjunto de datos.
7.2.3 ¡Manos a la obra!
Como ejemplo, vamos a utilizar parte del set de datos de la tesis doctoral “Empleo de descriptores fitoplanctonicos como biomonitores en la evaluacion de la calidad del agua en la costa del rıo de la Plata (Franja Costera Sur)” de M.B. Sathicq (2017) (Sathicq 2017), pública en el repositorio SEDICI de la UNLP.
7.2.3.1 Descargar los datos
Descargaremos dos sets de datos desde el repositorio del curso, y los debemos guardar en el Directorio de Trabajo del Proyecto que creamos para esta Unidad (ver cómo hacerlo en la Unidad 1).
Descargar el set de datos ambiental desde aquí: https://github.com/Limno-con-R/CILCAL2023/blob/main/datasets/environmental_data_OptimosPrime.csv
Descargar el set de dato de especies por muestras desde aquí: https://github.com/Limno-con-R/CILCAL2023/blob/main/datasets/species_data_OptimosPrime.csv
7.2.3.2 Instalar el paquete
Instalaremos el paquete optimos.prime (Sathicq, Nicolosi Gelis, and Cochero 2020) como se indica en la Unidad 1 y los cargaremos a nuestro espacio de trabajo actual:
library(optimos.prime)7.2.3.3 Cargar y previsualizar nuestro archivo de datos
Debemos cargar dos sets de datos. El primero contiene la información de las variables ambientales en cada sitio o muestra (“environmental_data_OptimosPrime”). El segundo contiene la información de abundancia relativa de los taxa en cada sitio o muestra (“species_data_OptimosPrime”).
env_data <- read.csv("environmental_data_OptimosPrime.csv") #Corroborar que el nombre del archivo coincida con el que vamos a utilizar, y que se encuentre en la carpeta de nuestro proyecto
species_data <- read.csv("species_data_OptimosPrime.csv") #Corroborar que el nombre del archivo coincida con el que vamos a utilizar, y que se encuentre en la carpeta de nuestro proyecto
#Alternativamente, para seleccionar un archivo con un cuadro de diálogo, se puede usar la función `file.choose()`, así:
env_data <- read.csv(file.choose()) #y seleccionar el CSV con los datos ambientales
species_data <- read.csv(file.choose()) #y seleccionar el CSV con los datos de abundancia de especies
#Cualquiera de estas dos opciones cargará nuestros datos a los objetos "env_data" y "species_data"Veamos brevemente cómo está organizada la información de nuestras especies:
str(env_data)env_data es un objeto data.frame con 11 obs. o filas (parámetros ambientales) y 51 variables o columnas (sitios muestreados, o muestras).
str(species_data)species_data es un objeto data.frame con 57 obs. o filas (especies) y 51 variables o columnas (sitios muestreados, o muestras).
¡Es importante que la cantidad de variables o columnas (sitios muestreados, o muestras) sea igual en ambos sets de datos!
7.2.3.4 ¡A la magia!
Vamos a ejecutar la función op_calculate() , que se encargará de realizar los análisis de óptimos ecológicos y rangos de tolerancia de los taxones que ingresamos en species_data a las variables ambientales que ingresamos en env_data . Y dispondremos de los resultados en un único objeto de tipo data.frame, que aquí lo llamaremos resultados.
resultados <- op_calculate(env_data, species_data)Cuando terminen los cálculos, en la consola nos indicará que ya podemos ver los resultados.
[1] "Optimum values and tolerance range calculated and placed in the final data frame"
[1] "Use View() to view data frame with results"
View(resultados)7.2.3.5 Los resultados
Si todo ha salido bien, debemos ver que el objeto resultados es una nueva matriz de 57 obs. o filas (especies) y ahora 34 variables o columnas (parámetros ambientales). Es decir, por cada uno de los parámetros ambientales que ingresamos (eran 11), vamos a tener una columna con su valor de óptimo ecológico y otras dos columnas con su rango ecológico máximo y su ecológico rango mínimo (33 columnas). La primera columna contendrá los nombres de las especies.
Veamos por ejemplo las columnas 2, 3 y 4.
La columna 2 (“Conductivity uS/cm”) indica el valor óptimo de conductividad eléctrica para las especies, y el nombre con las unidades fueron ingresadas por el usuario en la matriz de datos ambientales.
La columna 3 (“Conductivity uS/cm - HIGH”) indica el rango de tolerancia máximo, y la columna 4 (“Conductivity uS/cm - LOW”), el rango de tolerancia mínimo.
Es importante entender que los valores obtenidos responden a los óptimos, máximos y mínimos de los taxones en base a los valores de los parámetros ambientales ingresamos. Si luego incorporamos nuevas muestras (por ejemplo en otros ambientes) y encontramos las mismas especies en otras condiciones ambientales, los óptimos y rangos de tolerancia de esas especies van a cambiar. Es por eso que se les llama rangos ecológicos.
7.2.3.6 Graficar e interpretar nuestros resultados
El paquete optimos.prime nos puede generar gráficos de tipo caterpillar con nuestros resultados para cada variable ambiental, con la función op_plot(). A esta función le tenemos que indicar la matriz de resultados de la función op_calculate(), y le podemos indicar los argumentos label, para personalizar la etiqueta del gráfico, y/o el argumento html. Éste último indica si el gráfico producido se realizará en formato HTML, que permite interactuar con los datos usando el mouse, y es el valor predeterminado. Si html=FALSE, nuestro gráfico será una imagen plana.
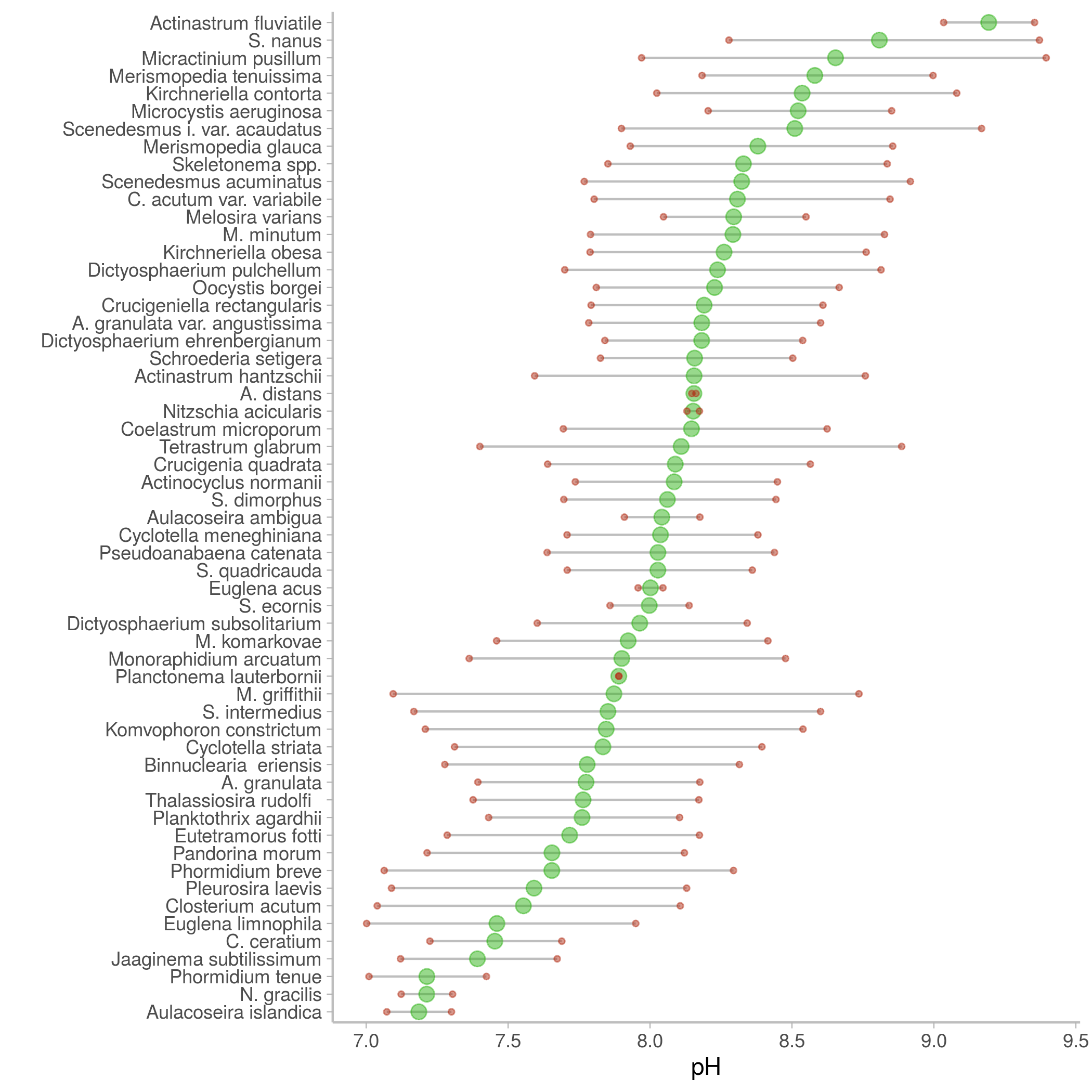
Figura 7.2. Óptimos ecológicos y rangos de variación para las especies de algas en referencia al pH.
En el gráfico, se verán las especies (en el eje Y) y la variable ambiental elegida (pH, en el eje X). Los puntos verdes indican el valor óptimo de cada especie, y los bigotes indican los rangos de tolerancia de cada especie a esa variable.
Aquellas especies con rangos de tolerancia más bajos (ejs. Actinastrum fluviale, o Euglena acus), son consideradas especies ‘sensibles’ a esa variable ambiental, y aquellas con grandes rangos de tolerancia (ejs. S.nanus o Tetrastrum glabrum) son consideradas especies ‘tolerantes’. A su vez en este ejemplo, aquellas con valores óptimos más altos (más arriba en el gráfico) son consideradas mas alcalófilas y las que se encuentran más abajo en el gráfico son especies más acidófilas. Es importante revisar aquellas especies que tienen rangos de tolerancia muy bajos (ej. A. distans), ya que puede ser el resultado de una baja cantidad de presencias de esa especie en las muestras.
op_plot(resultados)
#En la consola nos preguntará que variable queremos graficar, y deberemos ingresar el número de la variable. Por ejemplo, para seleccionar el pH escribiriamos el número 2.7.2.3.7 Algunas aclaraciones relevantes:
- ¿Nuestros datos no se encuentran en abundancia relativa?
Hay que indicarselo, cambiando el argumentoisRelAbaFALSE.
resultados <- op_calculate(isRelAb=FALSE)- ¿Nuestros datos ya fueron convertidos a log10?
Hay que indicarselo, cambiando el argumentoislog10aTRUE.
resultados <- op_calculate(islog10=TRUE)7.2.3.8 Shiny!
Hay una versión online para utilizar el paquete optimos.prime a través de un entorno Shiny, en:
https://limnolab.shinyapps.io/OptimosPrime-Shiny/